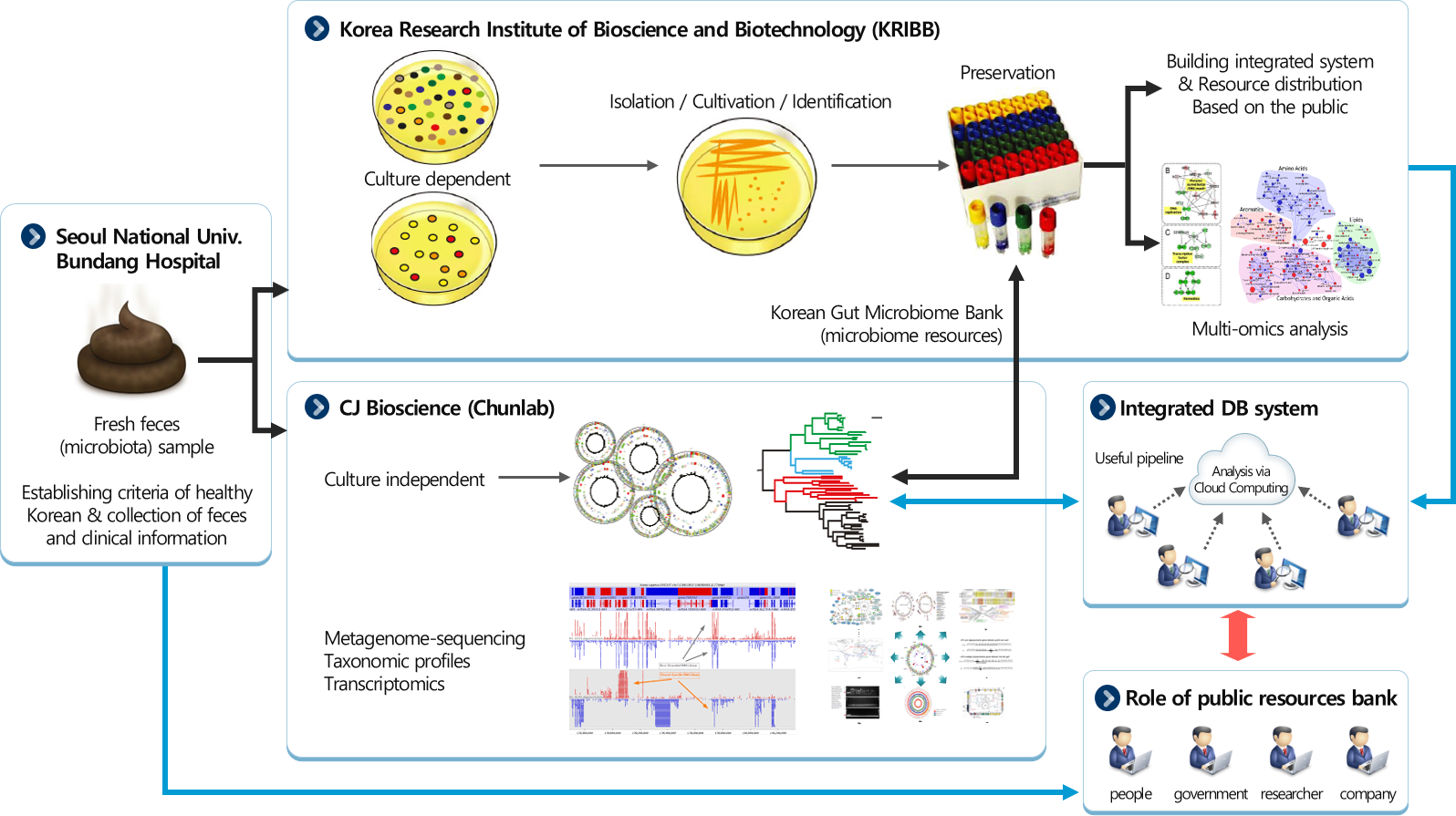
Korean Gut Microbiome Bank (KGMB) 프로젝트는 건강한 한국인의 장내 마이크로바이옴 메타게놈 분석을 기반으로, 생애주기별 주요 장내미생물들을 정의하고,
절대혐기성 미생물을 포함한 주요 장내 미생물들의 분리와 배양 최적화 기술 개발을 통해 세계 최고 수준의 마이크로바이옴 자원 뱅크 및 통합 정보 시스템을 구축함으로써 마이크로바이옴 자원 활용 및 실용화를 지원하고자 한다.
The purpose of the KGMB project is to define the major gut microbiome by life cycle based on the metagenome analysis of the gut microbiome of healthy Koreans, develop a technology for isolating and culturing major gut microbes,
including obligate anaerobic microorganisms, and build world-class microbiome resource bank and an integrated information system. We hope to accelerate the various utilization and commercialization of microbiome resources.
Through this, we intend to support the utilization and commercialization of microbiome resources.